引用本文: 陳振娜, 邵彥, 李筱榮. hsa-miRNA-451靶基因預測及生物信息學分析. 中華眼底病雜志, 2015, 31(6): 597-599. doi: 10.3760/cma.j.issn.1005-1015.2015.06.022 復制
微小RNA(miRNA)是一類長度約為19~25個核苷酸的非編碼小分子RNA,通過與靶信使RNA(mRNA)的特定序列互補或不完全互補結合,誘導靶mRNA剪切或阻止其翻譯[1],參與腫瘤、心血管疾病、代謝性疾病的細胞增生分化和細胞凋亡等多個生理過程并發揮調節作用[2]。hsa-miRNA-451位于染色體17q11.2,與原癌基因人類表皮生長因子受體2的17q11.2-21區域臨近[3],作為miRNA成員之一,具有高度的保守性,并已被證實參與乳腺癌、肝癌、非小細胞肺癌及糖尿病腎病等的發生發展[4-7]。有研究發現,miRNA在糖尿病視網膜病變(DR)的發生發展中發揮著重要作用[1, 8]。我們前期研究結果顯示,增生型DR(PDR)患者的增生膜活力與hsa-miRNA-451存在明顯相關性,其可能通過負反饋調節抑制增生膜的活力。但是其參與PDR的具體調控機制和生物學功能尚不完全清楚。為此,我們應用生物信息學方法對預測hsa-miRNA-451的靶基因進行了功能富集分析和信號轉導通路富集分析。現將結果報道如下。
1 材料和方法
選擇miRBase(http://www.mirbase.org)、miRanda(http://www.mirbase.org)、TargetScan(http://www.targetscan.org)、PicTar(http: //www.pictar.org)數據庫或預測軟件預測hsa-miRNA-451靶基因。選取上述3個及以上數據庫或預測軟件預測結果的交集作為進一步分析的基因集合。
利用基因本體(GO)[9]進行功能富集分類。通過注釋、可視化和整合發現的數據庫[10, 11]對預測的hsa-miRNA-451靶基因的基因集合進行基于GO的功能富集分析,以及基于京都基因與基因組百科全書的信號通路富集分析。采用Fisher精確概率法計算P值,以P<0.05為顯著性閾值;-Lg P值為P的負對數值,值越大代表富集越顯著。
2 結果
至少3個數據庫或預測軟件預測到hsa-miRNA-451靶基因的靶點18個(表 1)。
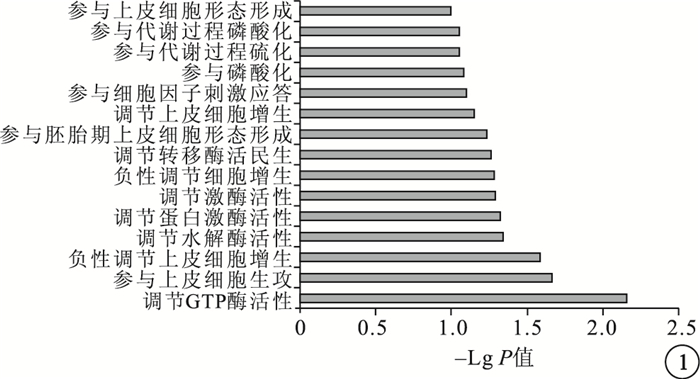
功能富集分析預測的結果顯示,hsa-miRNA-451調控的靶基因集合功能富集在調節上皮細胞發育、負性調節上皮細胞增生及多種酶活性等生物學過程(P<0.05)(圖 1)。
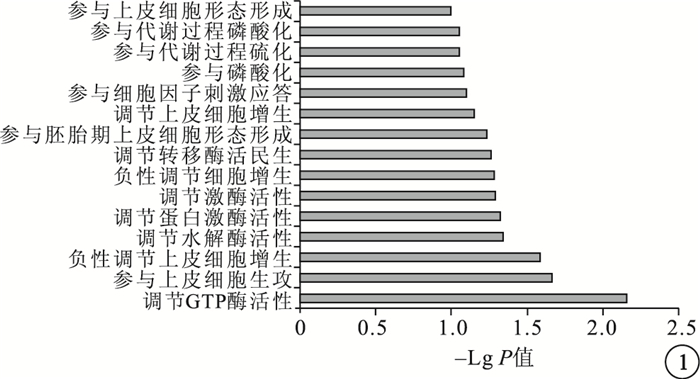 圖1
has-miRNA-451預測靶基因功能富集分析結果
圖1
has-miRNA-451預測靶基因功能富集分析結果
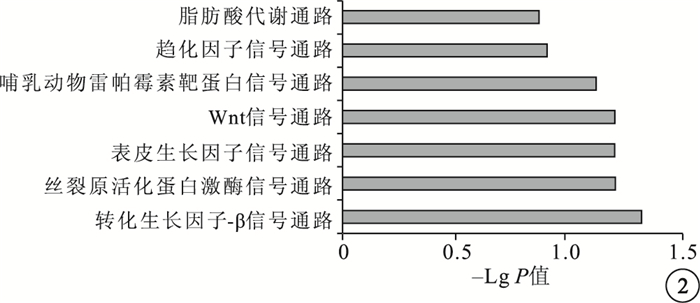
信號轉導通路富集分析預測的結果顯示,has-miRNA-451靶基因顯著富集于轉化生長因子-β信號通路、絲裂原活化蛋白激酶信號通路、表皮生長因子信號通路、Wnt信號通路、哺乳動物雷帕霉素靶蛋白信號通路、趨化因子信號通路、脂肪酸代謝通路(P<0.05)(圖 2)。
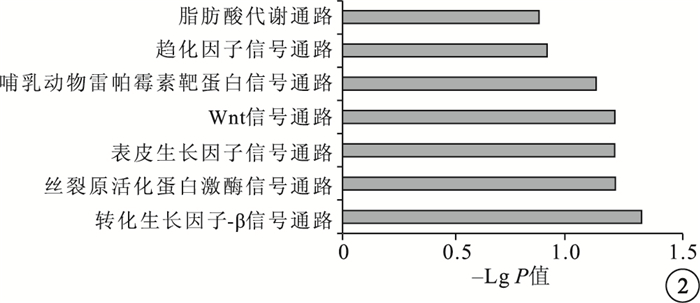 圖2
has-miRNA-451預測靶基因通路富集分析結果
圖2
has-miRNA-451預測靶基因通路富集分析結果
3 討論
在PDR發生過程中,視網膜色素上皮細胞等遷移到視網膜表面并增生形成支架,使得更多種類的細胞以此為基礎進行增生遷移[12],以此為切入點治療PDR正在成為新的研究方向。自Reinhart等[13]于2000年發現第1個具有轉錄后調節功能的小分子miR-let-7之后,miRNA已成為各個領域研究的熱點。miRNA在PDR發生、發展過程中的生物學作用也日益受到關注。在前期研究中,我們發現hsa-miRNA-451可抑制PDR增生膜的活力,因此擬對hsa-miRNA-451調控的潛在作用靶點及其在PDR中的具體作用及機制進行探究,為進一步研究提供依據。
生物信息學預測是尋找和發現靶基因的一種簡便方法,鑒于單一運用某種軟件會降低準確性,我們聯合應用了miRBase、miRanda、TargetScan和PicTar等幾種軟件進行靶基因功能富集分析和信號轉導通路富集分析。其中,miRBase為較早建立的miRNA靶標基因數據庫,較其他數據庫在功能和數據量方面更為全面,具有強大的綜合檢索能力,可以提供miRNA編號、序列信息、注釋、已經證實的預測基因靶標等信息[14, 15]。miRanda是第一代靶基因預測軟件,適用范圍廣,更新較快[16]。TargetScan同屬于第一代靶基因預測軟件,首次引入假陽性率預測靶基因預測結果。由于其要求種子序列能夠完全互補,因此預測的假陽性率較低,與實驗證實的靶基因吻合率較高[17, 18]。PicTar作為第二代靶基因預測軟件,兼顧了眾多第一代軟件的特點,將“完全匹配”和“不完全匹配”種子序列進行劃分并限制,可更好的降低假陽性率[19]。根據預測結果我們推測hsa-miRNA-451可能通過以上信號通路抑制上皮細胞增生,同時促進上皮細胞的發育發揮作用,這也與現有文獻支持轉化生長因子-β信號通路、絲裂原活化蛋白激酶信號通路等信號通路參與PDR的發生、發展[20-22]觀點相符。然而盡管我們采用多個軟件進行預測,通過取其交集部分來提高結果的可靠性,但是單純軟件預測的結果尚需進一步實驗結果證實。
我們通過生物信息學方法得到hsa-miRNA-451作用的靶點及其功能和信號傳導通路等結果,將以此為理論依據,同時結合實驗結果,進一步探討hsa-miRNA-451參與PDR的發病機制。
微小RNA(miRNA)是一類長度約為19~25個核苷酸的非編碼小分子RNA,通過與靶信使RNA(mRNA)的特定序列互補或不完全互補結合,誘導靶mRNA剪切或阻止其翻譯[1],參與腫瘤、心血管疾病、代謝性疾病的細胞增生分化和細胞凋亡等多個生理過程并發揮調節作用[2]。hsa-miRNA-451位于染色體17q11.2,與原癌基因人類表皮生長因子受體2的17q11.2-21區域臨近[3],作為miRNA成員之一,具有高度的保守性,并已被證實參與乳腺癌、肝癌、非小細胞肺癌及糖尿病腎病等的發生發展[4-7]。有研究發現,miRNA在糖尿病視網膜病變(DR)的發生發展中發揮著重要作用[1, 8]。我們前期研究結果顯示,增生型DR(PDR)患者的增生膜活力與hsa-miRNA-451存在明顯相關性,其可能通過負反饋調節抑制增生膜的活力。但是其參與PDR的具體調控機制和生物學功能尚不完全清楚。為此,我們應用生物信息學方法對預測hsa-miRNA-451的靶基因進行了功能富集分析和信號轉導通路富集分析。現將結果報道如下。
1 材料和方法
選擇miRBase(http://www.mirbase.org)、miRanda(http://www.mirbase.org)、TargetScan(http://www.targetscan.org)、PicTar(http: //www.pictar.org)數據庫或預測軟件預測hsa-miRNA-451靶基因。選取上述3個及以上數據庫或預測軟件預測結果的交集作為進一步分析的基因集合。
利用基因本體(GO)[9]進行功能富集分類。通過注釋、可視化和整合發現的數據庫[10, 11]對預測的hsa-miRNA-451靶基因的基因集合進行基于GO的功能富集分析,以及基于京都基因與基因組百科全書的信號通路富集分析。采用Fisher精確概率法計算P值,以P<0.05為顯著性閾值;-Lg P值為P的負對數值,值越大代表富集越顯著。
2 結果
至少3個數據庫或預測軟件預測到hsa-miRNA-451靶基因的靶點18個(表 1)。
功能富集分析預測的結果顯示,hsa-miRNA-451調控的靶基因集合功能富集在調節上皮細胞發育、負性調節上皮細胞增生及多種酶活性等生物學過程(P<0.05)(圖 1)。
 圖1
has-miRNA-451預測靶基因功能富集分析結果
圖1
has-miRNA-451預測靶基因功能富集分析結果
信號轉導通路富集分析預測的結果顯示,has-miRNA-451靶基因顯著富集于轉化生長因子-β信號通路、絲裂原活化蛋白激酶信號通路、表皮生長因子信號通路、Wnt信號通路、哺乳動物雷帕霉素靶蛋白信號通路、趨化因子信號通路、脂肪酸代謝通路(P<0.05)(圖 2)。
 圖2
has-miRNA-451預測靶基因通路富集分析結果
圖2
has-miRNA-451預測靶基因通路富集分析結果
3 討論
在PDR發生過程中,視網膜色素上皮細胞等遷移到視網膜表面并增生形成支架,使得更多種類的細胞以此為基礎進行增生遷移[12],以此為切入點治療PDR正在成為新的研究方向。自Reinhart等[13]于2000年發現第1個具有轉錄后調節功能的小分子miR-let-7之后,miRNA已成為各個領域研究的熱點。miRNA在PDR發生、發展過程中的生物學作用也日益受到關注。在前期研究中,我們發現hsa-miRNA-451可抑制PDR增生膜的活力,因此擬對hsa-miRNA-451調控的潛在作用靶點及其在PDR中的具體作用及機制進行探究,為進一步研究提供依據。
生物信息學預測是尋找和發現靶基因的一種簡便方法,鑒于單一運用某種軟件會降低準確性,我們聯合應用了miRBase、miRanda、TargetScan和PicTar等幾種軟件進行靶基因功能富集分析和信號轉導通路富集分析。其中,miRBase為較早建立的miRNA靶標基因數據庫,較其他數據庫在功能和數據量方面更為全面,具有強大的綜合檢索能力,可以提供miRNA編號、序列信息、注釋、已經證實的預測基因靶標等信息[14, 15]。miRanda是第一代靶基因預測軟件,適用范圍廣,更新較快[16]。TargetScan同屬于第一代靶基因預測軟件,首次引入假陽性率預測靶基因預測結果。由于其要求種子序列能夠完全互補,因此預測的假陽性率較低,與實驗證實的靶基因吻合率較高[17, 18]。PicTar作為第二代靶基因預測軟件,兼顧了眾多第一代軟件的特點,將“完全匹配”和“不完全匹配”種子序列進行劃分并限制,可更好的降低假陽性率[19]。根據預測結果我們推測hsa-miRNA-451可能通過以上信號通路抑制上皮細胞增生,同時促進上皮細胞的發育發揮作用,這也與現有文獻支持轉化生長因子-β信號通路、絲裂原活化蛋白激酶信號通路等信號通路參與PDR的發生、發展[20-22]觀點相符。然而盡管我們采用多個軟件進行預測,通過取其交集部分來提高結果的可靠性,但是單純軟件預測的結果尚需進一步實驗結果證實。
我們通過生物信息學方法得到hsa-miRNA-451作用的靶點及其功能和信號傳導通路等結果,將以此為理論依據,同時結合實驗結果,進一步探討hsa-miRNA-451參與PDR的發病機制。